
|
PIT00402 Какой компьютер в клетке? Мы с вами уже достаточно хорошо понимаем функции молекулярных "вычислений" в клетке, обеспечивающих все биологические взаимодействия с подобными клетками, а также с объектами других уровней организации. Конечно же это непознанная до конца система символьных преобразований нечисловых величин в режимах многозначных логик. Такая безадресная коллективная организация клякс и облаков лежит в основе любой живой системы от клетки до мозга. Такой подход к решению этой проблемы правильный, хотя очень сложный. Поэтому продолжаются попытки найти операции бинарной логики компьютеров в клеточных молекулах. Этот "легкий" путь, конечно же, ни к чему новому не приведет, лишь только каждый раз будет выявляться пропасть между тем, что удалось достичь в компьютерных моделях и что происходит на самом деле в клетке. Важно подчеркнуть, что на этом пути постоянно возникают символы биологической алгебры, часть из которых найдёт место в будущих формальных биологических моделях. Вот несколько самых последних работ. 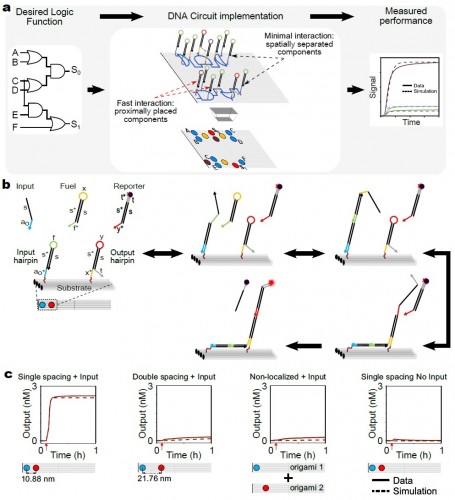 Рис. 1 [Chatterjee2017]. Пространственная организация управляет распространением сигнала. Биомедицинская синтетическая биология является развивающейся областью, в которой создаются клетки на инженерном генетическом уровне для выполнения новых функций [Gilad2017] 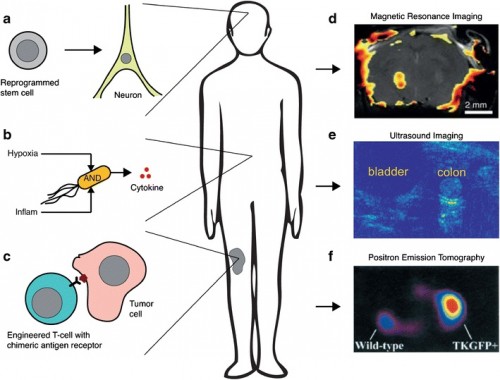 Рис.1 [Gilad2017]. Созидательный подход в биомедицинской синтетической биологии и молекулярной визуализации Статью [Chan2016] комментирует [Haynes2016], рисунок которого приведен ниже 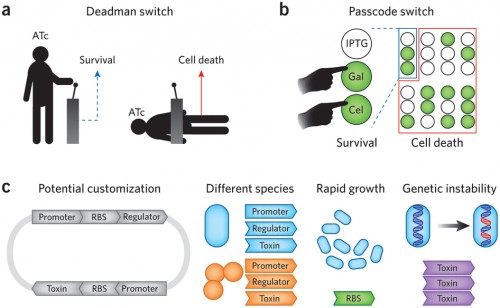 Рис. 1 [Haynes2016]. Генетические выключатели безопасности, построенные из взаимозаменяемых частей. Kim Caesar/Nature Publishing Group В работе [Lentini2017] показано, что искусственные клетки, которые способны воспринимать и синтезировать кворумные сигнальные молекулы, могут химически взаимодействовать с V. fischeri, V. harveyi, E. coli, and P. aeruginosa. 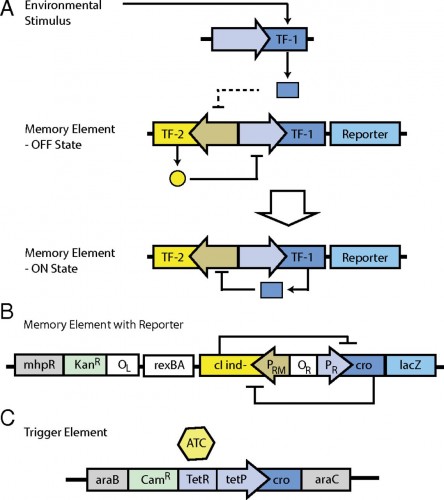 Рис. [Kotula2014]. Схема интегральной памяти {Сhatterjee2017} Gourab Chatterjee et al. A Spatially Localized Architecture for Fast and Modular Computation at the Molecular Scale // bioRxiv (2017), 110965. doi 10.1101/110965 @GeneMods 168 комментариев в Твиттере. Абстракт. Клетки используют пространственные ограничения для управления и ускорения потоков информации в ферментных каскадах и сигнальных сетях. Здесь мы покажем, что пространственная организация может быть мощный принципом конструирования для преодоления ограничений скорости и модульности в сконструированных молекулярных цепей. Мы создаем логические вентили и линии передачи сигналов пространственно организации реактивных шпилек ДНК на ДНК оригами. Распространение сигнала демонстрируется через линии передачи различной длины и ориентации, а также логические вентили модульно объединены в схемах, которые устанавливают универсальность нашего подхода. Поскольку реакции преимущественно происходят между соседями, идентичные шпильки ДНК могут быть использованы повторно через цепь. Коллективная (со)локализация элементов схемы уменьшается время вычислений с часов до минут по сравнению с цепями с диффундирующими компонентами. Подробные вычислительные модели позволяют создавать прогностическое схемное решение. Мы ожидаем, что наш подход будет стимулировать использование пространственных ограничений в области молекулярной инженерии в более широком смысле, в результате чего встроенные схемы молекулярного управления будут ближе к приложениям. {Gilad2017} Assaf A. Gilad, and Mikhail G. Shapiro. Molecular Imaging in Synthetic Biology, and Synthetic Biology in Molecular Imaging // Molecular Imaging and Biology (2017), 1-6. doi 10.1007/s11307-017-1062-1 {Lentini2017} Roberta Lentini et al. Two-Way Chemical Communication between Artificial and Natural Cells // ACS Central Science 3 (2), 117–123 (2017). DOI 10.1021/acscentsci.6b00330 {Halman2017} Justin R. Halman et al. Functionally-interdependent shape-switching nanoparticles with controllable properties // Nucleic Acids Research 45 (4), 2210-2220 (2017). doi 10.1093/nar/gkx008 {Chan2016} Clement TY Chan et al. 'Deadman'and'Passcode'microbial kill switches for bacterial containment // Nature chemical biology 12 (2), 82-86 (2016). doi 10.1038/nchembio.1979 Cited by 20 {Haynes2016} Karmella A. Haynes. Synthetic biology: Building genetic containment // Nature chemical biology 12 (2), 55-56 (2016). doi 10.1038/nchembio.2004 {Kotula2014} Jonathan W. Kotula et al. Programmable bacteria detect and record an environmental signal in the mammalian gut // Proceedings of the National Academy of Sciences 111 (13), 4838-4843 (2014). doi 10.1073/pnas.1321321111 Cited by 98 Более ранние ссылки можно найти в реферативной статье о сборке компьютеров внутри клеток годовой давности Peter Belobrov 02 Mar 2017 13:22
© International Open Laboratory for Advanced Science and Technology — MOLPIT, 2009–2026
|