
|
PIT00358 Микробиом глаз За 4 года это седьмой пост по поводу микробиома в ряду PIT00008, PIT00010, PIT00271, PIT00297, PIT00315 и PIT00349. Изменения микробиома глаз, связанные с ношением контактных линз, описаны в статье {Shin2016} Hakdong Shin, Kenneth Price, Luong Albert, Jack Dodick, Lisa Park, and Maria Gloria Dominguez-Bello. Changes in the eye microbiota associated with contact lens wearing // mBio 7 (2), e00198-16 (2016). doi 10.1128/mBio.00198-16 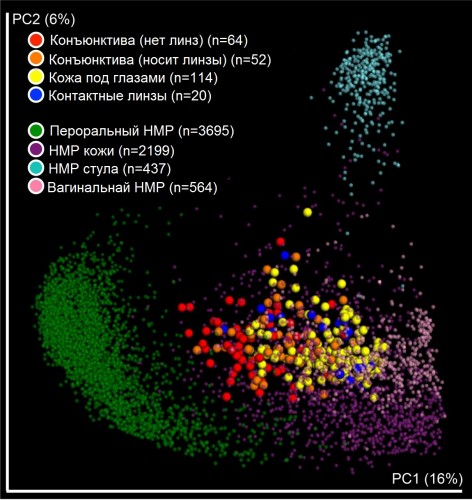 Рис.3 [Shin2016] Бета разнообразие в образцах конъюнктивы, кожи и контактных линз в сравнении с образцами из базы данных HMP (Human Microbiome Project = Проект микробиома человека). Участок PCoA был сгенерирован с использованием невзвешенных расстояний UniFrac. Маленькие точки (зеленый, фиолетовый, голубой и розовый) указывают на образцы из базы данных HMP и большие точки (красный, оранжевый, желтый и синий) показывают образцы этого исследования. Грамотное популярное объяснение того, почему контактные линзы могут изменить микробиом глаза написано в журнале BioTechniques 30/3/2016 Проект микробиома человека преподносит постоянно сюрпризы. Ниже список литературы из поста PIT00010 с добавлением числа цитирований на 24/11/2016 Y K Lee, S K Mazmanian. Has the Microbiota Played a Critical Role in the Evolution of the Adaptive Immune System? // Science 330 (6012), 1768-1773 (2010). Cited by 557 (# ) mib205 P R Hirsch & T H Mauchline. Who's who in the plant root microbiome? // Nature Biotechnology 30, 961–962 (2012). Cited by 38 (# ) mib206 D S Lundberg et al. Defining the core Arabidopsis thaliana root microbiome // Nature 488, 86–90 (2012). Cited by 455 (# ) mib207 D Bulgarelli et al. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota // Nature 488, 91–95 (2012). Cited by 380 (# ) mib208 A A Gust & T Nurnberger. Plant immunology: A life or death switch // Nature 486, 198–199 (2012). Cited by 13 (# ) mib209 Z Q Fu et al. NPR3 and NPR4 are receptors for the immune signal salicylic acid in plants // Nature 486, 228–232 (2012). Cited by 304 (# ) mib210 Дж Аккерман. Древнейшая социальная сеть // В мире науки, № 8, 10-17 (2012). J Ackerman. The Ultimate Social Network // Scientific American 306 (6), 36-43 (2012). Cited by 35 (# ) mib211 J Qin at al. A human gut microbial gene catalogue established by metagenomic sequencing // Nature 464, 59-65(2010). Cited by 4097 (# ) mib212 J V Weinstock. Autoimmunity: The worm returns // Nature 491, 183–185 (2012). Причины есть червей. Джоэл В. Вайншток объясняет как новые исследования с помощью кишечных паразитов указывают на потенциально новые методы лечения аутоиммунных заболеваний. Cited by 52 (# ) mib213 E A Grice and J A Segre. The Human Microbiome: Our Second Genome // Annual review of genomics and human genetics 13, 151-170 (2012). Cited by 121 (# ) mib214 The Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome // Nature 486, 207–214 (2012). Cited by 1997 (# ) mib216 The Human Microbiome Project Consortium. A framework for human microbiome research // Nature 486, 215–221 (2012). Cited by 681 (# ) mib217 J Shendure & E L Aiden. The expanding scope of DNA sequencing // Nature Biotechnology 30, 1084–1094 (2012). Cited by 190 (# ) mib218 S A Slavoff & A Saghatelian. Discovering ligand-receptor interactions // Nature Biotechnology 30, 959–961 (2012). Cited by 5 (# ) mib219 A P Frei et al. Direct identification of ligand-receptor interactions on living cells and tissues // Nature Biotechnology 30, 997–1001 (2012). Cited by 55 (# ) mib220 M Isalan. Systems biology: A cell in a computer // Nature 488, 40–41 (2012). Cited by 13 (# ) mib221 J R Karr at al. A Whole-Cell Computational Model Predicts Phenotype from Genotype // Cell, 150 (2), 389–401 (2012). Cited by 594 (# ) mib222 P L. Freddolino, S Tavazoie. The Dawn of Virtual Cell Biology // Cell, 150 (2), 248-250 (2012). Cited by 18 (# ) mib223 Peter Belobrov 24 Nov 2016 00:30
© International Open Laboratory for Advanced Science and Technology — MOLPIT, 2009–2026
|