
|
PIT00383 Хим и био полусинтетических организмов 23/2/2017 решил дополнить этот пост новыми ссылками и рисунками. После 15 лет напряженной работы, в лаборатории химической биологии и биофизики Ромесберга успешно спроектировали здоровый полусинтетический организм. Такая новая форма жизни может неопределенно долго хранить повышенной генетической информации, используя алфавит, состоящий из шести букв, которые образуют три пары оснований Janelle Weaver. Expanding the Genetic Alphabet to Create New Life Forms // BioTechniques 22/2/2017. Принципиально новый шаг для химической стабилизация неприродных нуклеотидтрифосфатов для расширения генетического алфавита в естественных условиях (in vivo) сделан а недавней работе [Feldman2017], графическая аннотация которой приведена ниже.  Аннотация статьи [Feldman2017]. Мы разработали неприродные пары оснований (UBP) и полусинтетический организм (SSO), который импортирует неприродные нуклеозидтрифосфаты и использует их для репликации ДНК, содержащей UBP. Тем не менее, распространение в UBP, по крайней мере частично, ограничено стабильностью неприродных трифосфатов, которые разлагаются под действием клеточных и секретируемых фосфатаз. Чтобы обойти эту проблему, мы теперь сообщают о синтезе и оценке неестественных трифосфатами с их Интересное продолжение работы [Ostrov2017], реферат которой сделан в PIT00310 (Что даёт замена 64 на 57 кодонов?), связано с разработкой полусинтетического организма на основе стабильного расширения генетического алфавита [Zhang2017]. Ниже приведены несколько абзацев из реферата Е Ефимова. Создан первый в мире жизнеспособный полусинтетический организм // vesti.ru 24/1/2017. Изначально спроектированные бактерии были достаточно слабыми и болезненными, в ходе экспериментов они и вовсе умирали. Происходило это по той причине, что микроорганизмам дополнительная пара оснований мешала, когда клетка начинала делиться. В течение следующих нескольких лет команда учёных разработала три метода проектирования новых "версий" бактерий E. coli, которые могли бы бесконечно удерживать свою новую пару оснований. Эти микроорганизмы могли жить нормальной и здоровой жизнью. Первым шагом на пути биологов стало создание улучшенной версии инструмента, который называется переносчик (транспортёр) нуклеотидов. Он вставляет в нужное место генетического кода синтетическую пару оснований. Был использован революционный инструмент редактирования генов CRISPR-Cas9 [Zhang2017], чтобы спроектировать бактерию кишечной палочки, которая не будет считать молекулы X и Y чужеродными "захватчиками". 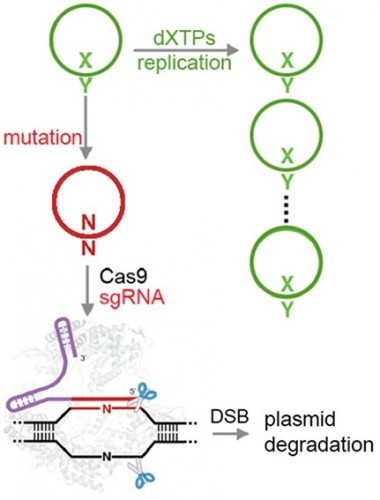 Рис.3а [Zhang2017] Подробнее получение полусинтетических организмов с расширенным генетического алфавита рассмотрено в обзоре [Malyshev2014]. Интересно, что такой подход полезен для поиска минимального бактериального генома [Martínez-García2016]. Ответ на наивный вопрос "а для чего это надо" только что пополнился нетривиальным ответом. Например, для предсказания мутационных эффектов из последовательности ковариаций [Hopf2017]. Думаю, и сама теория мутаций будет пересмотрена после анализа этих экспериментов. Не прошло и месяца, как появились новая работа [Steinegger2017], в которой предложены новая открытая программа выравнивания связанных кластеров (Linclust) MMseqs2.0 - ultra fast and sensitive search and clustering suite 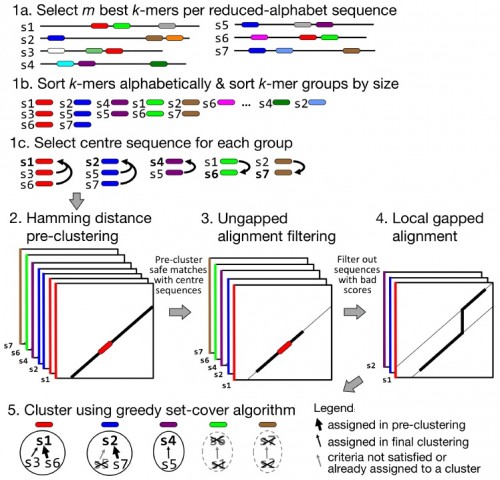 Рис. 1. [Steinegger2017] Пять этапов Linclust. Цветные окна представляют собой K-меры, выбранных в качестве потенциальных источников (затравок, семян) для расширения выравнивания в стадиях 2-4. {Malyshev2014} Denis A. Malyshev et al. A semi-synthetic organism with an expanded genetic alphabet // Nature 509 (7500), 385-388 (2014). doi 10.1038/nature13314 Cited by 161 {Ostrov2017} Nili Ostrov et al. Design, synthesis, and testing toward a 57-codon genome // Science 353 (6301), 819-822 (19 Aug 2016). DOI 10.1126/science.aaf3639 Cited by 14 {Martínez-García2016} Esteban Martínez-García, Víctor de Lorenzo. The quest for the minimal bacterial genome // Current Opinion in Biotechnology 42, 216-224 (2016). doi 10.1016/j.copbio.2016.09.001 {Hopf2017} TA Hopf et al. Mutation effects predicted from sequence co-variation // Nature Biotechnology 35, 128–135 (2017). doi 10.1038/nbt.3769 Cited by 3 {Zhang2017} Y Zhang et al. A semisynthetic organism engineered for the stable expansion of the genetic alphabet // PNAS 114 (6), 1317–1322 (2017). doi 10.1073/pnas.1616443114 Cited by 1 {Feldman2017} AW Feldman et al. Chemical Stabilization of Unnatural Nucleotide Triphosphates for the in Vivo Expansion of the Genetic Alphabet // Journal of the American Chemical Society 139 (6), 2464–2467 (2017). DOI 10.1021/jacs.6b12731 {Eisenstein2017} Michael Eisenstein. Pursuing the simple life // Nature Methods 14 (2), 117-121 (2017). doi 10.1038/nmeth.4158 {Starr2017} Tyler N. Starr, and Joseph W. Thornton. Exploring protein sequence-function landscapes // Nature Biotechnology 35 (2), 125-126 (2017). doi 10.1038/nbt.3786 N&V for [Hopf2017] {Steinegger2017} Martin Steinegger, and Johannes Soding. Linclust: clustering protein sequences in linear time // bioRxiv (2017), 104034. doi 10.1101/104034 [folder 701-170126-new64на57кодонов New papers] Peter Belobrov 27 Jan 2017 03:44
© International Open Laboratory for Advanced Science and Technology — MOLPIT, 2009–2026
|