
|
PIT00385 Гибрид человека и свиньи Такие «страсти-мордасти» наводят «тень на плетень». Невольно вспоминаешь пословицу «если человеку долго говорить, что он - свинья, то человек захрюкает». Реферат статьи [Wu2017] с обсуждением проблемы создания гибрида человека и свиньи Е. Ефимова. Создан гибрид человека и свиньи. Каковы преимущества и риски спорной методики? // vesti.ru 27/1/2017 позволил обратиться к оригинальным статьям [Mascetti2016, Wu2016, Wu2017]. Экспериментальные химеры широко признаны в качестве самых строгих тестов для проверки плюрипотентности стволовых клеток. Предимплантационная химеры обеспечивают клетки-доноры с доступом развития для всего плода и внеэмбриональной мезодермой (желтка, аллантоиса и амниотической мезодермы), тем самым обеспечивая широкую оценку потенциала развития донорских клеток. Тетраплоидные предимплантационная химеры, в частности, считаются наиболее полным испытанием плюрипотентности, потому что из стволовых клеток полученных полностью потомство мыши, что являются конечной точкой оценки [Mascetti2016]. Химеры являются монстрами воображения и давно установившимся исследовательским инструментом. Последние достижения, особенно те, которые касаются идентификации и генерации различных видов стволовых клеток, расширили репертуар и полезность межвидовых химер млекопитающих и привели к новым путям пониманию фундаментальной биологии и потенциальных клинических приложений [Wu2016]. Понятно, что на самом деле никакого гибрида человека и свиньи нет и не будет. Создана химера из плюрипотентных клеток человека и свиньи. Графическая аннотация ниже поясняет суть работы [Wu2017]. 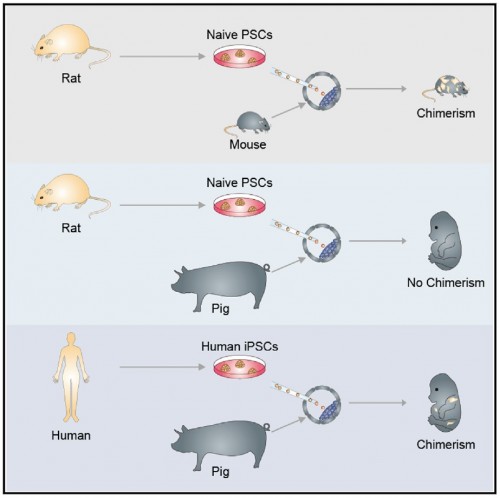 Человеческие плюрипотентные стволовые клетки хорошо приживаются в предимплантационные бластоцисты крупного рогатого скота и свиней, но показывают ограниченный химерный вклад в постимплантационные свиные эмбрионы. Это направление очень активно развивается для понимания биологических взаимодействий между клетками и тканями. Вот 3 самые последние работы для понимания состояния проблемы {Wu2017} Jun Wu et al. Interspecies Chimerism with Mammalian Pluripotent Stem Cells // Cell 168 (3), 473–486.e15 (2017). doi 10.1016/j.cell.2016.12.036 {Mascetti2016} Victoria L. Mascetti, Roger A. Pedersen. Contributions of mammalian chimeras to pluripotent stem cell research // Cell Stem Cell 19 (2), 163-175 (2016). doi 10.1016/j.stem.2016.07.018 {Wu2016} Jun Wu et al. Stem cells and interspecies chimaeras // Nature 540 (7631), 51-59 (2016). doi 10.1038/nature20573 Peter Belobrov 29 Jan 2017 12:00 23/2/2017 решил дополнить этот пост новыми ссылками и рисунками. После 15 лет напряженной работы, в лаборатории химической биологии и биофизики Ромесберга успешно спроектировали здоровый полусинтетический организм. Такая новая форма жизни может неопределенно долго хранить повышенной генетической информации, используя алфавит, состоящий из шести букв, которые образуют три пары оснований Janelle Weaver. Expanding the Genetic Alphabet to Create New Life Forms // BioTechniques 22/2/2017. Принципиально новый шаг для химической стабилизация неприродных нуклеотидтрифосфатов для расширения генетического алфавита в естественных условиях (in vivo) сделан а недавней работе [Feldman2017], графическая аннотация которой приведена ниже. 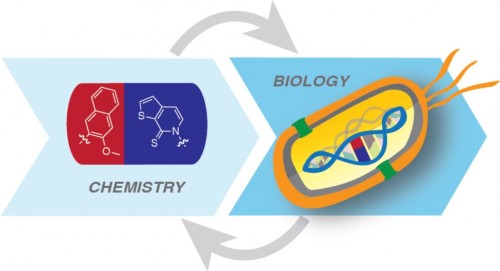 Аннотация статьи [Feldman2017]. Мы разработали неприродные пары оснований (UBP) и полусинтетический организм (SSO), который импортирует неприродные нуклеозидтрифосфаты и использует их для репликации ДНК, содержащей UBP. Тем не менее, распространение в UBP, по крайней мере частично, ограничено стабильностью неприродных трифосфатов, которые разлагаются под действием клеточных и секретируемых фосфатаз. Чтобы обойти эту проблему, мы теперь сообщают о синтезе и оценке неестественных трифосфатами с их Интересное продолжение работы [Ostrov2017], реферат которой сделан в PIT00310 (Что даёт замена 64 на 57 кодонов?), связано с разработкой полусинтетического организма на основе стабильного расширения генетического алфавита [Zhang2017]. Ниже приведены несколько абзацев из реферата Е Ефимова. Создан первый в мире жизнеспособный полусинтетический организм // vesti.ru 24/1/2017. Изначально спроектированные бактерии были достаточно слабыми и болезненными, в ходе экспериментов они и вовсе умирали. Происходило это по той причине, что микроорганизмам дополнительная пара оснований мешала, когда клетка начинала делиться. В течение следующих нескольких лет команда учёных разработала три метода проектирования новых "версий" бактерий E. coli, которые могли бы бесконечно удерживать свою новую пару оснований. Эти микроорганизмы могли жить нормальной и здоровой жизнью. Первым шагом на пути биологов стало создание улучшенной версии инструмента, который называется переносчик (транспортёр) нуклеотидов. Он вставляет в нужное место генетического кода синтетическую пару оснований. Был использован революционный инструмент редактирования генов CRISPR-Cas9 [Zhang2017], чтобы спроектировать бактерию кишечной палочки, которая не будет считать молекулы X и Y чужеродными "захватчиками". 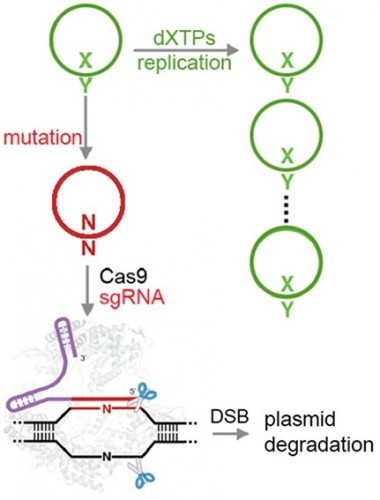 Рис.3а [Zhang2017] Подробнее получение полусинтетических организмов с расширенным генетического алфавита рассмотрено в обзоре [Malyshev2014]. Интересно, что такой подход полезен для поиска минимального бактериального генома [Martínez-García2016]. Ответ на наивный вопрос "а для чего это надо" только что пополнился нетривиальным ответом. Например, для предсказания мутационных эффектов из последовательности ковариаций [Hopf2017]. Думаю, и сама теория мутаций будет пересмотрена после анализа этих экспериментов. Не прошло и месяца, как появились новая работа [Steinegger2017], в которой предложены новая открытая программа выравнивания связанных кластеров (Linclust) MMseqs2.0 - ultra fast and sensitive search and clustering suite 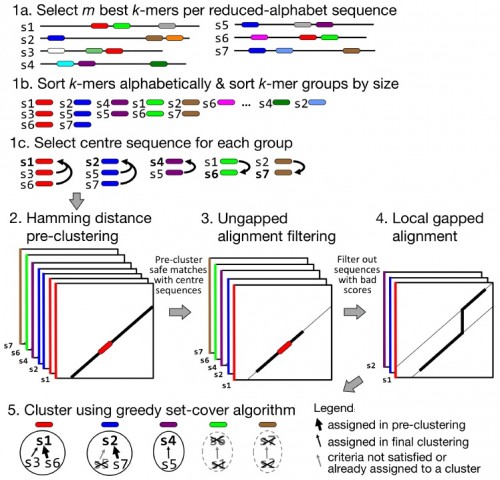 Рис. 1. [Steinegger2017] Пять этапов Linclust. Цветные окна представляют собой K-меры, выбранных в качестве потенциальных источников (затравок, семян) для расширения выравнивания в стадиях 2-4. {Malyshev2014} Denis A. Malyshev et al. A semi-synthetic organism with an expanded genetic alphabet // Nature 509 (7500), 385-388 (2014). doi 10.1038/nature13314 Cited by 161 {Ostrov2017} Nili Ostrov et al. Design, synthesis, and testing toward a 57-codon genome // Science 353 (6301), 819-822 (19 Aug 2016). DOI 10.1126/science.aaf3639 Cited by 14 {Martínez-García2016} Esteban Martínez-García, Víctor de Lorenzo. The quest for the minimal bacterial genome // Current Opinion in Biotechnology 42, 216-224 (2016). doi 10.1016/j.copbio.2016.09.001 {Hopf2017} TA Hopf et al. Mutation effects predicted from sequence co-variation // Nature Biotechnology 35, 128–135 (2017). doi 10.1038/nbt.3769 Cited by 3 {Zhang2017} Y Zhang et al. A semisynthetic organism engineered for the stable expansion of the genetic alphabet // PNAS 114 (6), 1317–1322 (2017). doi 10.1073/pnas.1616443114 Cited by 1 {Feldman2017} AW Feldman et al. Chemical Stabilization of Unnatural Nucleotide Triphosphates for the in Vivo Expansion of the Genetic Alphabet // Journal of the American Chemical Society 139 (6), 2464–2467 (2017). DOI 10.1021/jacs.6b12731 {Eisenstein2017} Michael Eisenstein. Pursuing the simple life // Nature Methods 14 (2), 117-121 (2017). doi 10.1038/nmeth.4158 {Starr2017} Tyler N. Starr, and Joseph W. Thornton. Exploring protein sequence-function landscapes // Nature Biotechnology 35 (2), 125-126 (2017). doi 10.1038/nbt.3786 N&V for [Hopf2017] {Steinegger2017} Martin Steinegger, and Johannes Soding. Linclust: clustering protein sequences in linear time // bioRxiv (2017), 104034. doi 10.1101/104034 [folder 701-170126-new64на57кодонов New papers] Peter Belobrov 27 Jan 2017 03:44 PIT00382 CRISPR и танцы молекул Вчера вместо Крещенского купания получил по почте новую удивительно интересную и заставляющую думать книгу Дениса Нобла "Танцы под дудочку жизни. Биологическая относительность". Denis Noble. "Dance to the tune of life. Biological relativity". Cambridge University Press. 2017 В этой книге формулируется теория биологической относительности, подчеркивающая, что живые организмы работают на разных уровнях сложности и, следовательно, должны быть проанализированы на многих масштабах с релятивистской точки зрения. Нобл объясняет, что все биологические процессы протекают в молекулярных, клеточных и организменных сетях. Интерактивный характер этих фундаментальных процессов лежит в основе биологической теории относительности, как таковой, и проблемы упрощенного молекулярного редукционизма. Нобл показывает, что такой интегративный подход возникает как необходимое следствие строгого применения математики к биологии. Опираясь на свою новаторскую работу в математической физике биологии, он показывает, что появление этого является глубоко гуманной картиной роли организма в сдерживании его химии, в том числе его генов, чтобы служить всему организму в целом, особенно при взаимодействии с его социальной средой. Эта гуманистический, целостный подход ставит под сомнение общепринятые представления о генах, которые находятся в центре, и которых придерживаются многие в современной биологии и в культуре. Денис Нобл является заслуженным профессором физиологии сердечно-сосудистой системы и директором центра вычислительной физиологии Оксфордского университета. Он является президентом Международного союза физиологических наук и членом Королевского общества, Великобритания. Краткое содержание книги "Танцы под дудочку жизни" 1 • Вселенная и принцип относительности 2 • Биологические масштабы и уровни 3 • Биологические сети 4 • Природа и происхождение клеток 5 • Слепой случай и естественный отбор 6 • Биологическая теория относительности 7 • Танцы нуклеотидов: природная генная инженерия 8 • Эпигенетика и релятивистская теория эволюции 9 • Относительность эпистемологии: Значение всего этого Смысл названия главы 9 напоминает слова Владимира Высоцкого "Ведь даже Эйнштейн - физический гений - весьма относительно всё понимал". Буду читать и пытаться найти переводчика, так как первая книга Дениса Нобла "Музыка жизни. Биология за пределами генома", вышедшая 10 лет назад, так и не была переведена на русский язык, хотя были сделаны переводы на многие языки мира. Подробнее см. на сайте musicoflife. Denis Noble. "The Music of Life. Biology beyond the Genome". 2006. 176 p. На нашем сайте более десятка постов основаны на "Музыке жизни". Проблемы природной генной инженерии связаны с танцами нуклеотидов (Гл.7) в книге "Танцы под дудочку жизни". Важным примером природной генной инженерии является CRISPR, см., например, {Mikuni2016} Takayasu Mikuni, Jun Nishiyama, Ye Sun, Naomi Kamasawa, and Ryohei Yasuda. High-throughput, high-resolution mapping of protein localization in mammalian brain by in vivo genome editing // Cell 165 (7), 1803-1817 (2016). doi 10.1016/j.cell.2016.04.044 Так как книга в России ещё мало доступна, рекомендую прочитать статью [Mikuni2016] вместе со статьёй [Noble2013], чтобы "навести на резкость" понимание современного взгляда на проблему молекулярных танцев под дудочку жизни в технологии CRISPR. {Noble2013} Denis Noble. A biological relativity view of the relationships between genomes and phenotypes // Progress in biophysics and molecular biology 111 (2), 59-65 (2013). doi 10.1016/j.pbiomolbio.2012.09.004 Peter Belobrov 20 Jan 2017 04:44 Отмечая 4 года посту PIT00003 P гранулы Вариант 2, 2 года - PIT00124 и год - PIT00207, хочу обратить ваше внимание на принципиально новый взгляд на комплексы РНК с белком. Этому мы обязаны в значительно степени лаборатории Дженнифер Додны (Jennifer A. Doudna), на 1-й странице сайта которой бактериальный иммунитет, РНК интерференция эукариот и контроль трансляции. Это, пожалуй, три основных инструмента книги клетки (CellBook), которые открыты в лаборатории Дженнифер. B.T. Staahl, M. Benekareddy, C. Coulon-Bainier, A. Ghosh, J.A. Doudna. Efficient protein-based genome editing by local delivery in the brain // Poster 317.09/T7 at 2016 Neuroscience Meeting, Session 317 - Neuroprotective Mechanisms: Signaling and Gene Expression of 14/11/2016. Abstract. RNA-guided genome editing triggered by the CRISPR-Cas9 DNA endonuclease has the potential to cure the underlying cause of a genetic disease. This therapeutic strategy will require tissue-specific delivery of the editing molecules in vivo, a goal that is particularly challenging in the brain. Here we show the Cas9 ribonucleoprotein (RNP) complexes can be used to edit post-mitotic neurons in the mouse brain. We also show that protein engineering of Cas9 increased the efficiency of in vivo neuronal cell editing ten-fold. Cas9 RNPs introduced locally in the brain trigger genome editing in cells including hippocampal, striatal and cortical neurons by a mechanism that involves spreading beyond the immediate site of injection. We also show that Cas9 RNP complexes are stable over 6 hours in blood plasma. These advances provide a robust technology for application of genome-editing in the brain to treat the underlying cause of neurological genetic diseases. Аннотация. РНК-управляемое редактирование генома инициируется CRISPR-Cas9 эндонуклеазами ДНК, которые имеет потенциал для лечения основной причины генетического заболевания. Эта терапевтическая стратегия требует доставки тканеспецифических молекул для редактирования in vivo. Эта цель является особенно сложной в головном мозге. Показано, что Cas9 рибонуклеопротеиновый (Cas9 RNP) комплексы могут быть использованы для редактирования пост-митотических нейронов в головном мозге мыши. Авторы также показали, что Cas9 белковая инженерия повысила эффективность редактирования нейронов in vivo в десять раз. Cas9 RNPs вводились локально для редактирования геномов переключающих клеток мозга, включая гиппокамп, полосатое тело и кортикальные нейроны по механизму, который включает в себя распространение за пределы непосредственного места инъекции. Авторы также показали, что Cas9 RNP комплексы стабильны в течение 6 ч в плазме крови. Эти достижения обеспечивают надежную технологию для применения редактирования генома в мозге при лечении основной причины неврологических генетических заболеваний. И хотя ни постера ни статьи нет ещё, вчера эти тезисы прокомментировала Kayt Sukel. A Safe New Approach for CRISPR-Cas9 Gene Therapy in Humans // BioTechniques 29/11/2016 Вышел хороший обзор Аннотация. TBA (редактирую) А пока почитаем про механизм работы CRISPR-Cas9, который на редкость хорошо и аккуратно описан в русскоязычной Википедии CRISPR. Интересный для понимания сути дела текст на сайте биомолекула.ру CRISPR-системы - иммунизация прокариот. Более подробное рассмотрение можно найти в самых первых больших обзорах Patrick D. Hsu, Eric S. Lander, and Feng Zhang. Development and applications of CRISPR-Cas9 for genome engineering // Cell 157 (6), 1262-1278 (2014). doi 10.1016/j.cell.2014.05.010 Cited by 1074 Jennifer A. Doudna, and Emmanuelle Charpentier. The new frontier of genome engineering with CRISPR-Cas9 // Science 346 (6213), 1258096 (2014).DOI 10.1126/science.1258096 Cited by 798 Peter Belobrov 30 Nov 2016 04:06 Интересная новость опубликована недавно David Cyranoski. CRISPR gene-editing tested in a person for the first time // Nature News, 15 November 2016. doi 10.1038/nature.2016.20988 Хороший перевод основной части этой статьи сделал Ефимов. Хотя в основе всех этих новостей сплетни о конкуренции генетиков Китая и США, развитие технологии CRISPR редактирования просто поражает! Peter Belobrov 20 Nov 2016 05:56 PIT00350 Геномное редактирование родословной Продолжаю пост PIT00290, в котором обсуждался эксперимент по трассировке родословной целостного организма комбинаторным и кумулятивным геномным редактированием. Вот точная ссылка на статью {McKenna2016} Aaron McKenna, Gregory Findlay, James A. Gagnon, Marshall Horwitz, Alexander Franz F. Schier, and Jay Shendure. Whole organism lineage tracing by combinatorial and cumulative genome editing // Science 353 (6298), 462-aaf7907 (2016). Аннотация DOI 10.1126/science.aaf7907 В отличие от препринта в статье есть подробная аннотация (такие только Science делает) с рис.1. 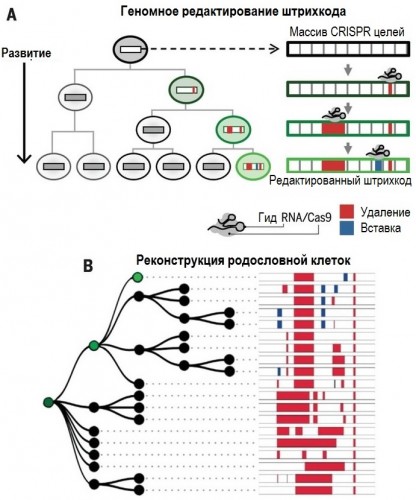 Рис. 1. Целостное восприятие результатов статьи [McKenna2016]. (А) Штрих-код CRISPR/cas9 целевых сайтов постепенно редактируется в течение многих клеточных делений. (B) Отредактированные последовательности штрих-кода связаны друг с другом на основе общих мутаций для восстановления деревьев клеточных родословных. Продвинулся в модели CellBook, обсуждение которой продолжу вскоре. Peter Belobrov 19 Oct 2016 00:00 Редактор метилирования ДНК уже не фантастика, а реальность. Для этого используют CRISPR/cas9, чтобы изменить состояние метилирования ДНК in vivo. Этот редактор продолжает развитие CRISPR экрана (см. PIT00347). Начнём с картинки в тексте Nicole Giese Rura 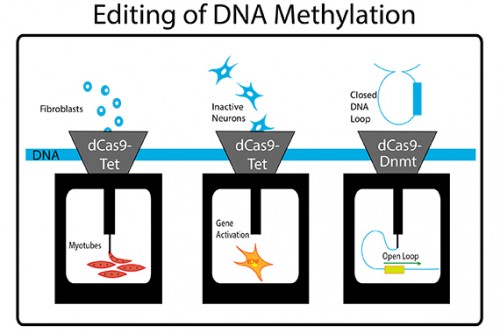 Принципиальная схема системы модификации метилирования при перепрограммировании клеток (слева), активирующих генов (в центре) и для изменения трехмерной структуры клеточной ДНК (справа). Это популярное для прессы изложение результатов статьи X. Shawn Liu, Hao Wu, Xiong Ji, Yonatan Stelzer, Xuebing Wu, Szymon Czauderna, Jian Shu, Daniel Dadon, Richard A. Young, and Rudolf Jaenisch. Editing DNA Methylation in the Mammalian Genome // Cell 167 (1), 233-247 (2016). doi 10.1016/j.cell.2016.08.056 с которой связаны следующие работы Angelo Amabile, Alessandro Migliara, Paola Capasso, Mauro Biffi, Davide Cittaro, Luigi Naldini, and Angelo Lombardo. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing // Cell 167 (1), 219-232 (2016). doi 10.1016/j.cell.2016.09.006 Yonatan Stelzer, Hao Wu, Yuelin Song, Chikdu S. Shivalila, Styliani Markoulaki, and Rudolf Jaenisch. Parent-of-origin DNA methylation dynamics during mouse development // Cell Reports 16 (12), 3167-3180 (2016). doi 10.1016/j.celrep.2016.08.066 и Yonatan Stelzer, Chikdu Shakti Shivalila, Frank Soldner, Styliani Markoulaki, and Rudolf Jaenisch. Tracing dynamic changes of DNA methylation at single-cell resolution // Cell 163 (1), 218-229 (2015). doi 10.1016/j.cell.2015.08.046 Peter Belobrov 18 Oct 2016 05:08 PIT00347 Некодирующий геном Рождение устойчивого понимания некодирующего генома произошло совсем недавно и определилось некодирующими белки РНК. И лишь сейчас изучение некодирующего генома становится систематическим (66% статей опубликованы за 4 последних года). Важное применение CRISPR (см. PIT00338) экран нашел для открытия функциональных некодирующих элементов путём опроса функциональных элементов в некодирующем геноме с высоким разрешением. По этой теме есть пара статей {Sanjana2016} Neville E. Sanjana, Jason Wright, Kaijie Zheng, Ophir Shalem, Pierre Fontanillas, Julia Joung, Christine Cheng, Aviv Regev, and Feng Zhang. High-resolution interrogation of functional elements in the noncoding genome // bioRxiv (2016), 049130. doi 10.1101/049130 {Wright2016} Jason B. Wright, and Neville E. Sanjana. CRISPR Screens to Discover Functional Noncoding Elements // Trends in Genetics 32 (9), 526-529 (2016). doi 10.1016/j.tig.2016.06.004 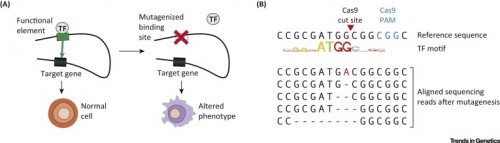 Рис.1. из [Wright2016] Нашлись эти статьи при поиске "тёмной материи" в клетке (соответствующий пост готовится). Вот аннотация статьи [Sanjana2016]. Наши результаты показывают, что cas9 опосредует систематическое рассечение некодирующих локусов, что может определить функциональные элементы, участвующие в регуляции генов. В сочетании с другими наборами данных и геномными анализами с высокой пропускной способностью, мы демонстрируем идентификацию областей, где изменения в контексте хроматина и связывание фактора транскрипции причинно связаны с потерей экспрессии генов. Обобщение этого подхода вместе с расширением объединенных экранов CRISPR в некодирующем геноме откроет новые пути обнаружения соответствующих элементов фенотипа и дополнительных методов для беспристрастного опроса «темной материи» генома и выяснения её значения в регуляции экспрессии генов. На русском языке на ленте.ру 18/5/16 А. Еникеев подробно реферирует статью [Sanjana2016] (правда, без ссылки на саму статью, но приводит из неё почти все рисунки) под названием Доковырялись Тайну «темной материи» генома разгадали с помощью CRISPR-Саs9. Среди 35 статей по запросу “Dark Matter” of the "noncoding genome" нашлась новая статья (глава из книги) с хорошим объяснением темной материи клетки. Samantha Barichievy, Loretta Magagula, Youtaro Shibayama, and Musa M. Mhlanga. Microbial Manipulation Host Dark Matter // Non-coding RNAs and Inter-kingdom Communication, pp. 27-52. Springer International Publishing, 2016. doi 10.1007/978-3-319-39496-1_2 Аннотация. В 2010 году Фрэнсис Коллинз назвал транскрибированные, но еще нетранслированные, компоненты человеческого генома «темной материей», термин, часто используемый астрофизиками для описания огромного количества невидимого гипотетического вещества, которое составляет большую часть нашей вселенной. С тех пор генетики многое установили в массиве биологических контекстах от рака до биологии развития. В последние годы быстро успехи были достигнуты в направлении выявления функциональной биологической роли длинных некодирующих РНК (lncRNAs), которые составляют 70-90% геномной темной материи млекопитающих. Становится все более очевидным , что основная функция нашего некодирующего генома состоит в регулировании кодирования генома. Это делает геномную темную материи привлекательной мишенью для эволюции патогенов, которым необходимо изменить среду клетки - хозяина в целях их выживания и распространения. В этом обзоре, мы обратили внимание на состав геномной темной материи млекопитающих, которыми манипулируют вирусные и микробные патогены. Мы также рассматриваем глубже участие нкРНК, в том числе усиливающих РНК (eRNAs), в формировании врожденного иммунного ответа против внутриклеточных патогенов. Этот комментарий подчеркивает, что как темный так и заумный наш некодирующий геном не разгадан до сих пор, особенно в контексте инфекционной биологии. Ana Lúcia Leitão, and Francisco J. Enguita. Non-coding RNAs and Inter-kingdom Communication. 2016. 251 p. Книгу можно назвать "Некодирующие РНК и биологические взаимодействия между царствами (Inter-kingdom Communication)". 14 глав разбиты на две знаменательные части Non-coding RNAs in Bacterial and Viral Interactions with Different Hosts Non-coding RNAs and Interactions Between Eukaryotic Organisms Peter Belobrov 16 Oct 2016 23:32 Техника, которая непосредственно активирует эндогенные гены и быстро преобразует один тип клеток в другой, преодолевая эпигенетические барьеры на пути превращения клеточных клонов фибробластов в нейроны, создана в работах [Treutlein2016, Black2016]. {Treutlein2016} Barbara Treutlein et al. Dissecting direct reprogramming from fibroblast to neuron using single-cell RNA-seq // Nature 534, 391–395 (2016). doi 10.1038/nature18323 {Black2016} Joshua B. Black at al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells // Cell Stem Cell 19 (3), 406-414 (2016). doi 10.1016/j.stem.2016.07.001 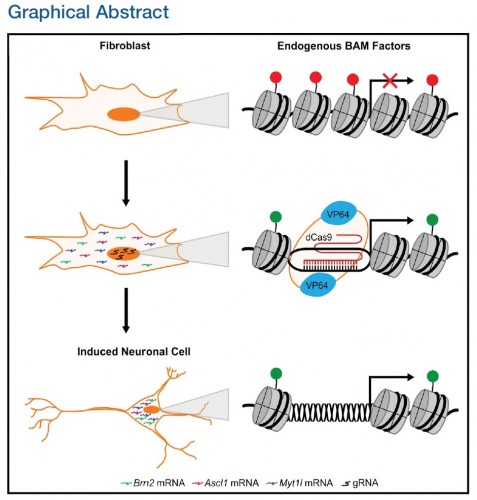 Превращение фибробластов в нейроны [Black2016]. Понятное объяснение этих результатов этих статей, их сравнение и описание важности вместе с результатами их предшественников сделано в работе [Zhou2016]. {Zhou2016} Yang Zhou, and Li Qian. Advanced Technologies Lead iNto New Reprogramming Routes // Cell Stem Cell 19 (3), 286-288 (2016). doi 10.1016/j.stem.2016.08.014 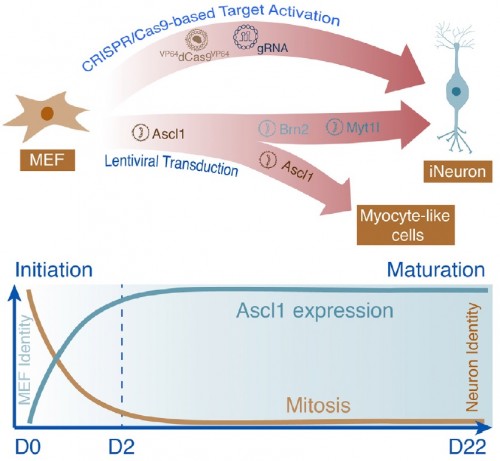 Рисунок 1. Сравнение молекулярных маршрутов перепрограммирования фибробластов в нейроны. Black и др. показал прямое перепрограммирование путем целенаправленной активации эндогенных факторов транскрипции с помощью CRISPR/Cas9 редактирования гена. Treutlein и его коллеги использовали одноклеточные РНК последовательности, чтобы реконструировать маршрут перепрограммирования, вызванного лентивирусной трансдукцией [Zhou2016]. Peter Belobrov 05 Oct 2016 00:02 PIT00328 Мультиплексная CRISPR система Исследования CRISPR/Cas9 системы (см PIT00311 и ссылки там) пополнились мультиплексной системой, позволяющих редактировать сразу несколько генов. Исследования сосредоточены на тонкой настройки CRISPR/cas9 системы для новой платформы. Приводом яаляется многоступенчатая стратегия последовательного включения одноцепочечных sgRNA кассет в одну плазмиду. Литература приведена в конце поста. Чтобы понимать работу CRISPR системы, посмотрим на рисунки с подписями, которые заимствованы из поста Emily Carlson 17/12/2015 How the CRISPR System Works 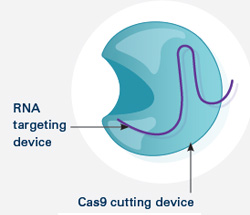 The CRISPR system has two components joined together • a finely tuned targeting device (a small strand of RNA programmed to look for a specific DNA sequence) • and a strong cutting device (an enzyme called Cas9 that can cut through a double strand of DNA). 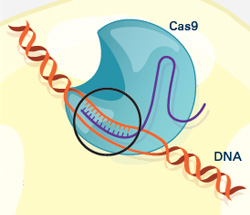 Once inserted into a cell, the CRISPR machine locates the target DNA sequence. CRISPR’s RNA recognizes and binds to the target DNA (circled in black). 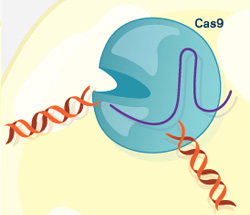 The Cas9 enzyme cuts both strands of the DNA. 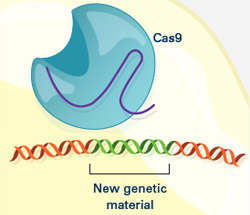 Researchers can introduce new genetic material, which the cell automatically incorporates into the gap when it repairs the broken DNA.  Among its many possible applications, the CRISPR system could be used to • Add a new gene that enables the organism to produce a pharmaceutical product (a biotechnology technique). • Help treat genetic diseases. • Create tailor-made model organisms to study human diseases. • Help produce replacements for damaged or diseased tissues and organs. References Cao, J., et al. “An easy and efficient inducible CRISPR/Cas9 platform with improved specificity for multiple gene targeting,” Nucleic Acids Research, 2016, DOI 10.1093/nar/gkw660. Dow, L., et al., “Inducible in vivo genome editing with CRISPR-Cas9,” Nature Biotechnology, 2015, doi 10.1038/nbt.3155. Kabadi, A., et al., “Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector,” Nucleic Acids Research, 2014, DOI 10.1093/nar/gku749. Vidigal, J. and Ventura, A. “Rapid and efficient one-step generation of paired gRNA CRISPR-Cas9 libraries,” Nature Communications, 2015, DOI 10.1038/ncomms9083. Peter Belobrov 15 Sep 2016 12:30 PIT00311 NgAgo детектив Исследования CRISPR/Cas9 системы (см. PIT00147, PIT00199, PIT00290 и особенно PIT00263) скорее всего будет отмечено Нобелевской премией в этом году. Поэтому, когда сегодня BioTechniques прислала письмо с темой "Будет ли новый метод NgAgo лучше, чем CRISPR?", обратился к этой детективной истории. Nathan Blow. To Edit or Not - The NgAgo Story  В мае 2016 года исследователи из университета Hebei сообщили в журнале Nature Biotechnology, что Natronobacterium gregoryi Argonaute (NgAgo) может функционировать как ДНК-зависимая эндонуклеаза, способная редактировать геном человека [{Gao2016}]. Результаты были мощными, так как NgAgo, в отличие от Cas9, похоже, не надо смежного с фотоспэйсером мотива (РАМ = protospacer-adjacent motif), что делает его удобным для целенаправленного редактирования генома.  Эта картинка из галереи рисунков NIH поясняет РАМ. Посмотрите в Гугле Images для NgAgo, 94 сообщения и 12027 просмотров NgAgo в https://groups.google.com/forum/#!forum/crispr и {Gao2016} Feng Gao, Xiao Z. Shen, Feng Jiang, Yongqiang Wu, and Chunyu Han. DNA-guided genome editing using the Natronobacterium gregoryi Argonaute // Nature biotechnology 34, 768–773 (2016). doi 10.1038/nbt.3547 Peter Belobrov 23 Aug 2016 13:34 PIT00290 CellBook - эксперимент Мне трудно даже было представить, что экспериментальное исследование родословной клеток начнётся так быстро. Поэтому, когда на сайте vesti.ru встретил новость На "пленку" ДНК записали историю жизни всех клеток, то был в шоке. Стал проверять - мало ли что напишут журналисты, у которых генетический код меняется при каждом делении (к сожалению, некоторые студенты так думают, принимая геном за генетический код). Но когда прочитал об использовании биоштрихкода при изучении родословной клеток, то понял, что "лёд тронулся, господа присяжные заседатели". Замечу здесь, что на этом сайте понятие родословной клетки используется давно, т.к. с моей точки зрение понятие клеточной линии (lineage) не позволяет грамотно описать биологию развития организмов, когда сетевой и стволовой принципы при симметричном и ассиметричном делении перемешиваются. Надо строить точную модель родословной! Вот препринт этой статьи Удивительно, что c этой новостью русские вести опередили BioTechniques на 3 дня, хотя статья Jeffrey M. Perkel. Tracing Cell Lineages with CRISPR/Cas // BioTechniques 3/6/2016 более сдержанная. Модель CellBook надо доводить "до ума"! Peter Belobrov 31 May 2016 13:14 PIT00263 Адаптивный иммунитет прокариот Как говорится, "кто прозевал - тот опоздал". Так и с направлением CRISPR - адаптивного иммунитета прокариот. Научных статей уже в 2016 ~ 1220, всего ~ 139000. Дело даже не в количестве, а том, что врождённый и адаптивный иммунитет есть у всех участников симбиозов. А причём тут Е.В. Кунин и йогурт? Читайте сами статью, рисунок из которой приведен ниже
https://www.quantamagazine.org/20150206-crispr-dna-editor-bacteria/ 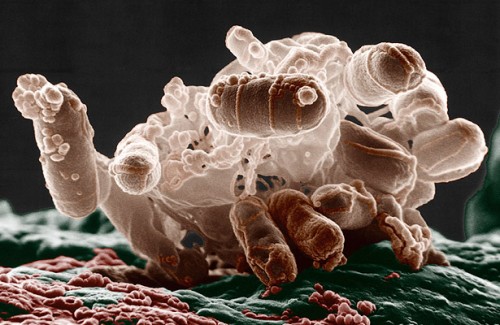 Photo by Eric Erbe, Colorization by Christopher Pooley. USDA. Микробы, такие как E. coli может использовать CRISPR в качестве оружия в их миллионо-летний борьбы с вирусами. Кому лень читать на английском, так хотя бы на русском посмотрите по диагонали (не пожалеете!) это http://biomolecula.ru/content/1498 Статья Ирены Артамоновой. CRISPR-системы: иммунизация прокариот (14 ноября, 2014 г.) CRISPR — это система специфического иммунитета прокариот, характерная как для бактерий, так и для архей. CRISPR-структуры впервые были описаны почти 30 лет назад, однако их функция долгое время оставалась загадкой. CRISPR-системы состоят из геномных кассет, в которые записывается информация о вирусных или плазмидных инвазиях, и Cas-белков, обеспечивающих молекулярный механизм иммунитета. В ответ на инфекцию клетка с CRISPR вырезает из чужеродного генома небольшой фрагмент и встраивает его в кассету. Благодаря такому механизму роста, CRISPR-системы являются уникальным примером эволюции «по Ламарку». Высокоэффективное узнавание ДНК, лежащее в основе работы CRISPR, оказалось привлекательно и для практического использования, и сейчас CRISPR-системы служат для точных манипуляций с самыми различными геномами, в том числе и с геномом человека. https://ru.wikipedia.org/wiki/CRISPR Короткие палиндромные повторы, регулярно расположенные группами (англ. CRISPR — Clustered Regularly Interspaced Short Palindromic Repeats) — это прямые повторы и разделяющие их уникальные последовательности в ДНК бактерий и архей, которые совместно с ассоциированными генами (Cas, англ. CRISPR-associated genes) обеспечивают защиту клетки от чужеродных генетических элементов (бактериофагов, плазмид). CRISPR-кассеты обнаружены в геномах многих бактерий и большинства архей. Повторы имеют длину от 24 до 48 пар нуклеотидов; они имеют бивалентную симметрию (англ. dyad symmetry), но, как правило, не являются истинными палиндромами. Повторы разделены вариабельными участками ДНК, спейсерами, примерно одинаковой длины. Спейсеры соответствуют по нуклеотидной последовательности определённым фрагментам ДНК чужеродных генетических элементов (протоспейсерам). В связи с этим было предположено и затем показано, что последовательности, разделяющие повторы, происходят из последовательностей геномов бактериофагов, и, соответственно, обеспечивают защиту клеток от инфекций. https://ru.wikipedia.org/wiki/Cas9 Cas9 (англ. CRISPR associated protein 9, CRISPR-ассоциированный белок) — это управляемая при помощи РНК-гидов эндонуклеаза, связанная с адаптивной иммунной системой CRISPR у ряда бактерий Peter Belobrov 26 Jan 2016 09:12 PIT00199 Редактирование генома При чтении словосочетания "Gene Editing", которое чаще всего означает "редактирование генома" невольно возникает вопрос "а редактор кто и что и как он редактирует?" Редактирование генома, или редактирование генома инженерными нуклеазами (GEEN = ГРИН) - тип генной инженерии, в которой ДНК вставляется, заменяется или удаляется из генома с помощью искусственно сконструированных нуклеаз, или "молекулярных ножниц" (см. PIT00183). После двух встреч (вместе более 10 часов) с Л.И. Патрушевым, который руководит группой анализа и коррекции генома в лаборатории биотехнологии ИБХ РАН, идея реализации полииндикаторных чипов (пличипов) стала реальностью, которую я буду обсуждать на семинаре сегодня. Детальное изучение геномов растений, животных и человека открывает широчайшие возможности применения полученных знаний в биотехнологии и медицине. Однако только данных о нуклеотидных последовательностях геномов недостаточно для понимания функциональных взаимосвязей отдельных элементов геномов и их роли в формировании фенотипических признаков и патогенезе отдельных заболеваний. В постгеномную эпоху активно развиваются методы, позволяющие манипулировать с ДНК в геномах, а также визуализировать и управлять экспрессией генов и работой регуляторных элементов. Тем не менее далеко не все методы отвечают высоким требованиям к их эффективности, безопасности и доступности для широкого круга исследователей. В последние несколько лет появились новейшие методы редактирования геномов это системы TALEN (Transcription Activator-Like Effector Nucleases) и CRISPR (Clustered Regulatory Interspaced Short Palindromic Repeats)/Cas9. Эти появившиеся относительно недавно системы уже зарекомендовали себя как эффективные и надежные инструменты геномной инженерии. Данный обзор в основном посвящен применению указанных систем для редактирования геномов основных модельных объектов современной биологии, а также для функционального скрининга геномов, создания клеточных моделей наследственных заболеваний человека, изучения эпигеномов и визуализации клеточных процессов. Кроме того, рассмотрены основные методы конструирования подобных систем, проанализировано их действие, обсуждаются некоторые проблемы, которые ожидают исследователей при применении этих инструментов редактирования геномов. Это аннотация обзорной статьи Вопрос в начале поста пока открыт, думаю. Peter Belobrov 03 Apr 2015 01:56 Редактирование генома факторами транскрипции, такими как нуклеазы с доменами типа "цинковые пальцы" {zinc finger nucleases (ZFN)}, активаторами транскрипции типа эффекторных нуклеаз {transcription activator like effector nucleases (TALEN)}, или CRISPR/Cas9 системой позволяет точно и эффективно редактировать гены в эмбрионах, особенно, если им дать хороший шок, как это сделано недавно в работе T Kaneko et al. Simple knockout by electroporation of engineered endonucleases into intact rat embryos 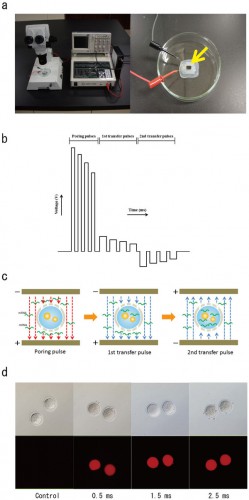 (a) Суперэлектропоратор НЕПА 21 (слева) и стеклянная камера с металлическими пластинами (справа). Эмбрионы помещали в линию между металлическими пластинами (стрелка). (b) Иллюстрация электрических импульсов электропоратора. (c) первый сканирующий импульс высокого напряжения и короткой продолжительности делает микроотверстия в зоны прозрачности (слева) и мРНК в PBS затем перемещается в цитоплазму с несколькими начальными импульсами низкого напряжени и большой длительности после углубившкго импульса (в центре). МРНК затем переносили в ооциты следующими импульсами измененной полярности (справа). (d) Флуоресцентный анализ меченого тетраметилродамином декстрана, который был введен в эмбрионы путем электропорации импульсами шириной 0, 0,5, 1,5, и 2,5 мс. Peter Belobrov 21 Oct 2014 02:14
© International Open Laboratory for Advanced Science and Technology — MOLPIT, 2009–2026
|